Bernd sitzt zusammen mit Dr. Hajo Kries und spricht über das Design von Naturstoffen. Hajo ist Wissenschaftler am Hans-Knöll-Institut (HKI), dem Leibniz-Institut für Naturstoff-Forschung und Infektionsbiologie in Jena und leitet dort die Nachwuchsgruppe biosynthetisches Design von Naturstoffen.

Die beiden sprechen über die Proteinsynthese in Zellen bzw. Mikroogranismen und wie man diese ändern muss, um neue Naturstoffe produzieren zu können. Hajo hat hier vor allem die Nichtribosomale Peptidsynthetasen (NRPS) im Blick, mit deren Hilfe er bisher unbekannte Peptide erzeugen möchte, die als Wirkstoffe verwendet werden können. Dabei verändert er den genetischen Code der Nichtribosomalen Peptidsyntheasen (NRPS) mit Hilfe gerichteter Evolution solange, bis das von Ihm ausgewählte Peptid von der NRPS erzeugt wird.
(Im Podcast gibt es Kapitelmarken, die den Zwischenüberschriften hier im Text entsprechen, so dass es einfacher ist, bestimmte Teile erneut zu hören. Nicht jede Kapitelmarke hat eine Zwischenüberschrift, manchmal fassen wir mehrere Kapitel zusammen.)
Was bedeutet biosynthetisches Design von Naturstoffen?
In der Natur gibt es viele wichtige Wirk- oder Giftstoffe, die zum Beispiel aus Pflanzen, Pilzen oder Mikroben erzeugt werden. Früher hat man diese Substanzen vor allem durch Isolation bzw. Extraktion gewonnen. Bei dem biosynthetischen Design von Naturstoffen nutzt man Werkzeuge der synthetischen Biologie, also Methoden aus der Mikrobiologie und Biotechnologie, um in Mikroorganismen gezielt neue Wirkstoffe herzustellen.
-
- Penicilline – Wikipedia Artikel
- Alexander Fleming – Wikipedia Artikel
- Synthetische Biologie – Wikipedia Artikel
- Molekularbiologie – Wikipedia Artikel
- Mikrobiologie – Wikipedia Artikel
- Biotechnologie – Wikipedia Artikel
Welche Naturstoffe werden designt?
Beispiele für solche Naturstoffe sind Polyketide wie z.B. Erythromycin, ein Makrolid Antibiotikum. Des Weiteren gibt es bestimmte Terpene wie Artemisinin gegen Malaria oder Taxol als Krebsmedikament. Hajos Gruppe ist allerdings spezialisiert auf nicht ribosomale Peptide und Polyketide.
Ziel des biosynthetischen Designs
Optimierung der Struktur des Naturstoffs im Hinblick auf:
-
- Wirkungen /Nebenwirkungen.
- Erhöhung der Ausbeute des Naturstoffs
- Entwicklung neuer Wirkstoffe auf Basis der natürlichen Bausteine durch neues „Zusammensetzen“
Designwerkzeuge
-
- Peptid Synthese– Wikipedia Artikel
- Nicht ribosomale Peptidsynthetase (NRPS) – Wikipedia Artikel
Was ist eine Nichtribosomale Petidsythetase (NRPS)?
Hajo führt aus, dass es sowohl einen ribosomalen Weg als auch einen nichtribosomalen Weg gibt, um Proteine zu erzeugen. Der Weg über das Ribosom stellt den Standardweg der Protein- bzw. Peptidsynthese in den meisten Lebewesen dar und ist evolutionär schon sehr alt.
Ribosomen bestehen aus Nukleinsäuren; diese Nukleinsäuren des Ribosoms bauen aus Aminosäuren die Proteine zusammen. Dabei wird DNS im Zellkern der Zellen in mRNS übersetzt. Dies wird aus dem Zellkern der Zellen ausgeschleusst und gelangt im Cytosol an das Ribosom. Das Ribosom nutzt diese mRNS als Bauplan, um die darin kodierten Proteine zu bauen.
Des Weiteren erwähnt Hajo die Ribosomalen Posttranslational-modifizierten Peptide (RIPP).
Drei Wege der Proteinsythese:
-
- Ribosom – Wikipedia Artikel
- Nichtribosomales Peptid – Wikipedia Artikel
- RIPP – Wikipedia Artikel (englisch)
Aufbau der NRPS
Eine NRPS ist aufgebaut wie ein Fließband aus Proteinmodulen, jedes Modul ist für den Einbau eines Bausteins (z.B. Aminosäure) zuständig. Vorteil des Modulaufbaus ist, dass relativ einfach gezielte Modifikationen möglich sind; so kann ein Organismus neben den proteinogenen Aminosäuren auch andere Bausteine in den gewünschten Wirkstoff einbauen.
Kodierung der NRPS Module
Die Peptidsequenz ist kodiert im NRPS Gen, nach der Herstellung NRPS Proteins steht das Ergebnis Peptid bereits fest. Daraus folgt, dass die NRPS nicht programmierbar ist.
Das Ribosom hat die mRNA als Vorlage, um ein Protein zu machen. Die mRNA entscheidet, welches Protein vom Ribosom produziert wird, das Ribosom ist also programmierbar.
Ein Vorteil der NRPS ist, dass NRPS mehrere hundert unterschiedliche Bausteine in die Synthese einarbeiten kann, u.a. können neben Aminosäuren auch nicht-proteinogene oder nicht natürliche Bausteine eingebaut werden. Beispiel Synthese von Daptomycin.
Diese strukturelle Flexibilität lässt Raum für den Einbau gewünschter Modifikationen wie fluoreszenter Bausteine oder Bausteine, mit denen man ein Peptid mit anderen Molekülen verlinken (zusammenfügen) kann. Voraussetzung sind Amino- und/oder Säuregruppen, um die Bausteine in das Peptid integrieren zu können. So ist die Startergruppe bei dem erwähnten Daptomycin eine Fettsäure.
Modifikation der NRPS und gerichtete Evolution
Um das von der NRPS erzeugte Peptid zu verändern, muss Hajo die NRPS, also das vorgefertigte Fließband modifizieren. Dabei verfolgt Hajo unterschiedliche Strategien wie:
-
- Austausch bzw. Rekombination von natürlichen Modulen in anderer Reihenfolge
- Veränderung einzelner Module und Einbau des veränderten Moduls
Für die Veränderung der Module muss der genetische Code eines Moduls geändert werden; hierfür nutzt Hajo Techniken der gerichteten Evolution
„…. Gerichtete Evolution funktioniert sehr ähnlich wie die Evolution der Natur, aber mit einem Ziel. Die natürliche Evolution passiert ja einfach ohne dass irgend etwas oder irgend jemand vorgibt, wohin es gehen soll! …“- Zitat Hajo.
Für die Technik der gerichteten Evolution hat 2018 Francis Arnold unter anderem den Nobelpreis Chemie bekommen. Sie hat gezeigt, dass man auf diesem Weg nützliche Biokatalysatoren herstellen kann.
Die Größe eines durchschnittlichen Moduls ist ca. 50kDalton. 1 Aminosäure entspricht dabei ca. 100Dalton. Das bedeutet, dass ein Modul aus ca. 500 Aminosäuren besteht. Da die NRPS aus mehreren Modulen bestehen, können diese leicht aus mehreren tausend Aminosäuren aufgebaut sein, die dann auch noch korrekt gefaltet werden müssen.
-
-
Structures of a dimodular nonribosomal peptide synthetase reveal conformational flexibility – By Science, Wissenschaftlicher Artikel zu Strukturaufklärung NRPS (open access)
-
Wer sich zu Röntgenkristallographie informieren möchte. Wir haben dazu schon eine Episode im Wirkstoffradio: WSR011 Proteinstrukturen aufklären mit der Röntgenkristallographie – Interview mit Dr. Yvette Roske
-
- Pressemitteilung zur Veränderung der NRPS für Click-Chemie.
- Click-Chemie – Wikipedia Artikel
Design enzymatischer Domänen eines Moduls
Eine Domäne ist ein Teil innerhalb des NRPS Moduls, die eine spezifische Funktion übernimmt, z.B. stellt die Domäne eine Bindetasche mit katalytischer Funktion dar. In der Regel bilden drei Domänen ein Modul. Ziel von Hajos Arbeit ist es, Domänen so zu verändern, dass neue Bausteine dort verarbeitet werden können. Hajo führt dies an unterschiedlichen Beispielen aus.
-
- Hier ein ausführlicher Review zum Aufbau der NRPS und der Domänen: Nicht‐ribosomale Peptidsynthese – Prinzipien und Perspektiven, leider befindet sich der Artikel hinter einer Paywall, ist also nur für diejenigen zugänglich, die ein Abo der entsprechenden Fachzeitschrift haben. Daher werden wir nicht direkt auf den Artikel verlinken, sondern stellen euch den“Digital Object Identifier (DOI)” zur Verfügung: 10.1002/ange.201609079
Analyse der gerichteten Evolution mittels MS
-
- Massenspektrometrie (MS) – Wikipedia Artikel
Bei der MS wird ein Molekül anhand seiner Masse analysiert. Dabei kann das Molekül auch innerhalb des MS fragmentiert (zerkleinert) werden. Da Moleküle nach bestimmten Mustern zerfallen, ergeben die Fragmente eine Art Fingerabdruck. Dieser ist dann für eine bestimmte Aminosäure bzw. ein ganzes Peptid spezifisch, so dass man damit komplizierte organische Moleküle identifizieren kann.
Zur Analyse der NRPS wird die Synthese bereits bei dem Einbau der Aminosäuren unterbrochen und mittels MS die Zusammensetzung des synthetisierten Peptids bestimmt.
Hajo führt aus, dass sein MS so empfindlich ist, dass er eine Messerspitze Kokain, die in einem Olympischen Schwimmbecken aufgelöst wurde, noch nachweisen könnte.

-
- Wildtyp – Wikipedia Artikel
- Mutagenese – Wikipedia Artikel
Gramicidin S
Hajos Modellpeptid ist Gramicidin S .
-
- Gramicidine – Wikipedia Artikel
- Gramicidin S – Wikipedia Artikel
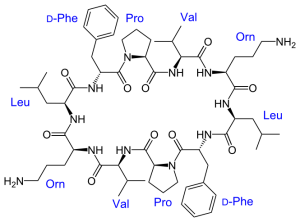
Anwendung der modifizierten NRPS-Module
Ziel ist ein perfekt designter Wirkstoff, der sich biologisch herstellen lässt.
Beispiel Penicillin Biosythese
Gewinnung aus dem Penicillium Pilz. Die verschiedenen Penicilline wie z.B. Ampicillin werden dann durch Modifizierung auf chemisch biotechnologischem Wege hergestellt.
-
- Ampicillin – Wikipedia Artikel
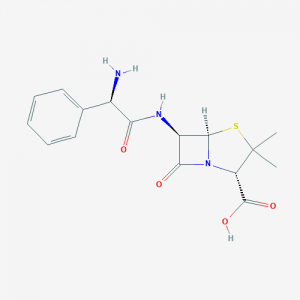
Effektivität und Rekombination der NRPS-Module
Hajo beschreibt, dass sie bei der Modifizierung von Modulen schon gute Erfolge erzielt haben und andere Aminosäuren mit hoher Spezifität einsetzen können. Für die Rekombination der modifizierten Module verwendet Hajos Team DNA Moleküle, um die Position der Module zu definieren.
-
- Prof. Dr. Helge B. Bode– Gruppe an der Goethe Universität Frankfurt am Main
Hajos Werdegang
-
- Prof. Donald Hilvert an der ETH Zürich.
- beta-peptide – Wikipedia Artikel
- Hajos Gruppe am HKI
Lieblingsmolekül
Hajos Lieblingsmolekül ist HG3.17 sein erstes Enzym, das er mittels gerichteter Evolution verändert hat. Die Effizienz des Enzyms als Biokatalysator konnte durch diese Veränderungen um mehr als 100-fach gesteigert werden.
-
- Pressemitteilung der ETH (englisch)
- Der primär Artikel zu dem Molekül : “Precision is essential for efficient catalysis in an evolved Kemp eliminase.” befindet sich leider hinter einer Paywall, ist also nur für diejenigen zugänglich, die ein Abo der entsprechenden Fachzeitschrift haben. Daher werden wir nicht direkt auf den Artikel verlinken, sondern stellen euch den“Digital Object Identifier (DOI)” zur Verfügung: 10.1038/nature12623
Miriam Agler-Rosenbaum hat bei uns in der Episode: WSR034 Wirkstoffproduktion: Von der Flasche im Labor bis zum Tank in der Industrie schon über den Betrieb eines Biotechnikums gesprochen.
Wir bedanken uns ganz herzlich bei Dr. Hajo Kries für die Zeit und seine Erläuterungen und Ausführungen zu seinem Forschungsgebiet.
Wir freuen uns immer über Feedback: per Mail unter info@wirkstoffradio.de, in den Kommentaren unter den einzelnen Episoden, über Twitter @wirkstoffradio oder auch als Bewertung bei iTunes oder panoptikum.social.
Wirkstoffradio-Feedback-Telefon +49 (0)30 746 910 64
Wirkstoffradio ist lizenziert unter einer Creative Commons Namensnennung - Keine Bearbeitungen 4.0 International Lizenz.